Tercer Milenio
En colaboración con ITA
Epidemiología digital: rastreando virus por internet
Cuando buscamos en internet los síntomas de una gripe o de la covid, y también cuando hablamos de nuestra salud en las redes sociales, dejamos un rastro de datos que puede ser utilizado por los epidemiólogos para identificar el brote de un virus emergente o un nuevo foco de una epidemia.

A finales de 2019, dos sistemas de monitoreo digital de enfermedades, HealthMap y ProMED, detectaron los primeros signos de alarma procedentes de la ciudad de Wuhan, en China, días antes de que lo advirtiera la Organización Mundial de la Salud (OMS). El equipo de Health Map obtuvo pistas a partir de una nota de prensa publicada en internet y ProMED detectó que se estaban produciendo conversaciones en Weibo, una red social China, sobre una neumonía de origen desconocido.
Los datos procedentes de los motores de búsqueda, de las redes sociales, chats y otras publicaciones en la red son una suerte de "migas digitales" para los epidemiólogos, en palabras de John Brownstein, uno de los fundadores de HealthMap, profesor de Medicina de Harvard y jefe de Innovación del Boston Children’s Hospital. Constituyen una cantidad ingente de datos, que proporciona pistas a diario sobre los brotes de distintas enfermedades infecciosas u otros problemas de salud.
En diciembre de 2019, dieciséis días antes del anuncio del brote de SARS-CoV-2 por parte de las autoridades locales de Wuhan, entre los posts y búsquedas realizados en WeChat, una popular aplicación china de mensajería utilizada por mil millones de personas cada mes, empezó a aparecer con más frecuencia la palabra ‘feidian’, que en chino mandarín significa síndrome respiratorio agudo grave. Hasta entonces, los usuarios de WeChat no habían escrito la palabra ‘feidian’ demasiadas veces, pero entre el 15 y el 29 de diciembre su uso aumentó y lo hizo de forma especialmente rápida el 30 de diciembre, un día antes de que el brote de una neumonía atípica se hiciera público.
Este fenómeno fue analizado de forma retrospectiva por investigadores del Hospital de Xi’an en China, analizando los datos de WeChat Index, un servicio de acceso público que proporciona la frecuencia con la que los usuarios de esta aplicación escriben determinadas palabras. Los investigadores concluyeron que utilizando esta herramienta, y ‘feidian’ como palabra clave, se podría haber detectado el primer brote de covid dos semanas antes. También identificaron un incremento en el uso de términos como ‘SARS’, ‘coronavirus’, ‘nuevo coronavirus’, ‘dificultad para respirar’, ‘disnea’ o ‘diarrea’, aunque estas palabras clave no funcionaban igual de bien para detectar la epidemia de forma anticipada.
Dos semanas de desfase
Los sistemas de vigilancia epidemiológica son esenciales para identificar brotes de nuevos virus emergentes o de enfermedades ya conocidas y permitir a los países tomar medidas frente a situaciones que pueden derivar en epidemias o incluso en una pandemia como la actual. También lo son para seguir la dinámica de infecciones estacionales como la gripe, que se repiten año tras año. Tradicionalmente estos sistemas de vigilancia epidemiológica trabajan con los datos clínicos y microbiológicos que les proporcionan los hospitales y los laboratorios que analizan muestras de pacientes en busca de patógenos. Pero, ya sea por falta de recursos a nivel local o por otros factores, se ha estimado que habitualmente existe un desfase de una a dos semanas desde que se produce un brote hasta que lo comunica un organismo oficial como el Centro Europeo para el Control y Prevención de Enfermedades (o sus equivalentes americanos o chinos). Con el fin de acortar este desfase temporal, los expertos en análisis de datos hace más de una década que han puesto su mirada en internet. En este tiempo se han desarrollado iniciativas como Google Flu Trends que tenía como objetivo predecir las epidemias de gripe a partir de las búsquedas realizadas en Google.
Sistemas de información como HealthMap (creado en 2006) y ProMED (creado en 1994), integran grandes cantidades de datos que emplean para monitorizar los brotes de enfermedades infecciosas y para proporcionar información a tiempo real destinada tanto a las agencias de salud pública locales, a la OMS o a los CDC como al gran público. Pero en general, todavía no emplean información procedente de las redes sociales, a no ser que sea de fuentes expertas en salud pública bien identificadas. Por ejemplo, HealthMap, analiza y filtra información procedente de noticias publicadas en la red y de fuentes gubernamentales. También se nutre de proyectos de ciencia ciudadana como Flu Near You y Outbreaks Near Me, en las que millones de usuarios, aportan de forma desinteresada sus síntomas de gripe o de covid, o los resultados de sus test, para poder rastrear de forma colaborativa los brotes de estas infecciones.
Aunque todavía nos encontramos en los albores de la epidemiología digital, la gran cantidad de datos y la rapidez con que estos se pueden transmitir y analizar, será de gran utilidad para anticiparnos y rastrear futuras epidemias, como ya empezamos a ver. Aun así, existen una serie de limitaciones que habrá que tener en cuenta. Entre ellas, las principales son la fiabilidad y precisión de los datos y los modelos generados, como se vio en el caso de Google Flu Trends, y cuestiones relativas a la privacidad y seguridad de los usuarios que contribuyen y proporcionan sus datos.
Fiabilidad y límites
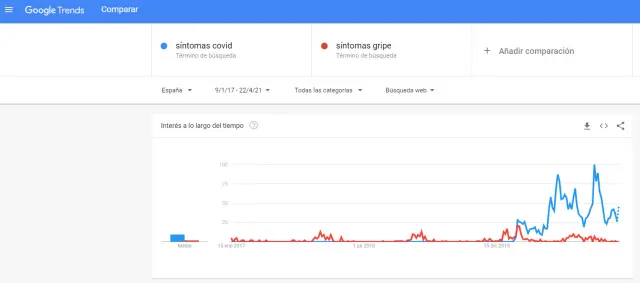
Para que los modelos basados en los buscadores de internet sean fiables, estos deben testear conjuntos de palabras clave para elegir aquellas que mejor representan la evolución de los datos reales. Por ejemplo, el economista y profesor de la Universidad de Navarra, Francesc Pujol, en un reciente artículo publicado en su blog, ha empleado la herramienta de acceso público Google Trends (Tendencias de búsquedas de Google), que muestra la evolución de los términos de búsqueda más populares en un periodo de tiempo concreto, para visualizar si existen correlaciones entre las búsquedas y los casos de gripe o covid. En su análisis, Pujol emplea de forma aislada términos simples como ‘gripe’, ‘covid’, o ‘síntomas covid’, y encuentra una serie de correlaciones ilustrativas con las olas de gripe, en el pasado, o de covid, a partir de 2020. Estas no dejan de ser curiosidades que, como se puso de manifiesto con el modelo mucho más complejo de Google Flu Trends (basado en 45 frases clave), la realidad tarde o temprano puede terminar por desmentir.
Por ejemplo, algunas predicciones pueden sobreestimar el número de casos porque los usuarios buscan información en la red sobre la enfermedad después de que se haya hablado de ella en las noticias, y no porque realmente tengan síntomas. En otras situaciones la explicación es más divertida; como en 2007, cuando en Estados Unidos se produjo un pico de búsquedas en Google de la palabra ‘cólera’ no por un brote de la enfermedad, sino porque la presentadora de televisión Oprah Winfrey había recomendado la novela de Gabriel García Márquez ‘El amor en los tiempos del cólera’ en su club de lectura.
Así pues, para evitar estas confusiones, serán necesarios modelos más fiables que empleen algoritmos de inteligencia artificial que vayan aprendiendo a partir de grandes cantidades de datos y se vayan adaptando a las dinámicas reales que intentan predecir, que son complejas. HealthMap ya los emplea y, a partir de una base de datos de millones de artículos que describen brotes de infecciones reales etiquetados y categorizados por su equipo de investigadores, su sistema aprende a distinguir la información útil de la que es espuria.
Otra limitación es que la información recabada no sea representativa del conjunto de la población. En función del buscador o de la red social utilizados, se puede estar dejando fuera de los modelos a grupos de personas por razón de su edad, sexo, idioma o nivel socioeconómico, entre otros, y, por ello, los modelos que se deriven estarán condenados al fracaso. En determinadas redes sociales se ha visto que la mayoría de contribuciones provienen únicamente de una fracción pequeña de gente, alrededor del 10%, que representa a los usuarios más activos. Las redes sociales también son fuente de noticias falsas y de bulos que pueden introducir ruido de fondo a los modelos predictivos.
Otro problema es el riesgo para la privacidad de los usuarios, por ejemplo cuando se emplean los datos de geolocalización de los teléfonos móviles para el rastreo de casos y contactos en una epidemia o se usan las ‘migas digitales’ que dejamos en nuestros paseos por la red. Deberá garantizarse el uso agregado y anónimo de estos datos y que se preservan los derechos de los usuarios, entre ellos su privacidad.
Google Flu Trends: auge y declive
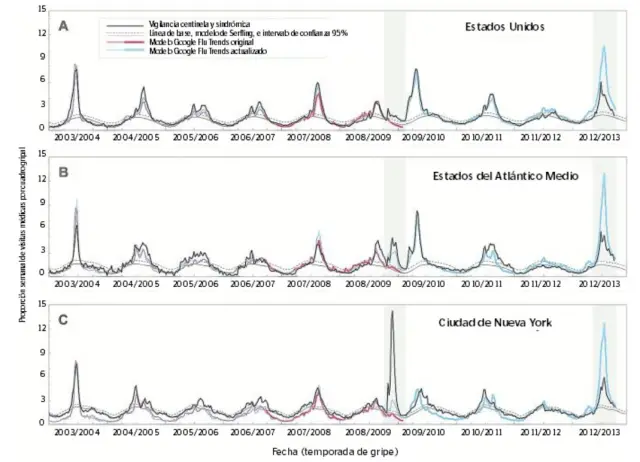
En 2008, Google creó Google Flu Trends un servicio que proporcionaba predicciones sobre la incidencia de la gripe basadas en los datos introducidos en su buscador. Para crear su modelo predictivo, Google escogió 50 millones de combinaciones potenciales de términos relacionados con la gripe y analizó la frecuencia con la que eran utilizadas, en el periodo 2003-2006, en relación con la incidencia de gripe. De estas, se seleccionaron las 45 frases que mejor se ajustaban a la evolución de los casos de gripe y se testearon con los datos del siguiente año. El modelo precedía la incidencia de gripe casi a la perfección y lo hacía mejor que los modelos basados en los datos de los Centros para el Control y Prevención de Enfermedades de los Estados Unidos (CDC).
Pero en 2009 cambiaron las tornas. Entre 2003 y 2008 las epidemias de gripe habían seguido unos patrones estacionales estrictos, mientras que en 2009 los primeros casos en Estados Unidos empezaron a detectarse en abril y se transformaron en una pandemia de proporciones globales (la de la gripe A producida por la cepa H1N1). En este escenario, el modelo de Google falló estrepitosamente: no se ajustaba a la realidad, ya que había subestimado con creces el número de casos.
Aprendiendo de sus errores, el equipo de Google recalibró el modelo, pero este volvió a fallar en 2012, esta vez sobreestimando el número de casos de gripe en el estado de Nueva York. De hecho, un estudio publicado en ‘PLoS Computational Biology’ que comparó las predicciones de Google Flu Trends con la evolución real de la gripe entre 2003 y 2013, encontró errores sustanciales tanto en las estimaciones temporales como de intensidad de las epidemias. En 2015, Google dejó de publicar sus predicciones y actualmente solo facilita sus datos para fines de investigación. Aún así, Flu Trends fue uno de los precursores en el empleo de los datos de la red para vigilancia epidemiológica.
Múltiples datos
Los modelos epidemiológicos basados en inteligencia colectiva serán probablemente un complemento y no un sustituto de los sistemas tradicionales de vigilancia epidemiológica, y beberán de múltiples fuentes. De nuestro comportamiento en las redes, pero también de las contribuciones voluntarias en proyectos de ciencia colaborativa en los que se reportan los síntomas de infecciones o se comunica la presencia en una zona de vectores de enfermedades transmisibles, como por ejemplo los mosquitos. Otra fuente de datos a tener en cuenta son los accesorios conectados a internet que miden constantes vitales como la frecuencia cardíaca o la temperatura y que pueden reflejar a tiempo real los síntomas de infecciones como la gripe o la covid. La Universidad del Estado de Oregon y la empresa Kinsa, han distribuido más de un millón de termómetros conectados a la red y trabajan en un sistema para poder predecir la evolución de estas enfermedades. En el caso de la gripe afirman que su algoritmo predice los incrementos de casos dos semanas antes que las predicciones de los CDC.
-Ir al suplemento Tercer Milenio
Apúntate y recibe cada semana en tu correo la newsletter de Tercer Milenio