Tercer Milenio
En colaboración con ITA
Covid-19
Fotografía genómica de la pandemia en España
Un solo genotipo del virus generó el 60% de los casos a inicios de marzo y otro, que tuvo su origen durante el verano en Aragón, se ha extendido por España y domina la segunda ola de la pandemia en toda Europa.

¿Podemos saber cómo se inició en febrero-marzo la epidemia de covid-19 en España, uno de los países europeos más afectados? ¿Cuántas veces entró el virus a nuestro país y cómo se propagó durante la primavera? La respuesta a algunas de estas preguntas la tiene el estudio comparativo de miles de genomas del SARS-CoV-2, el virus causante de la covid-19, procedentes de pacientes infectados durante la primera ola de la pandemia.
Un estudio como este, para entender cuáles fueron los primeros pasos del coronavirus en España, es el que ha llevado a cabo el consorcio de secuenciación SeqCovid, que acaba de publicar un informe de la fase inicial del proyecto, en el que se describe y analiza la diversidad genómica del SARS-CoV-2 durante los primeros tres meses de la epidemia, de febrero a abril.
El consorcio SeqCovid aúna el trabajo de más de treinta hospitales de toda España, de laboratorios de genómica y de especialistas en salud pública, con el objetivo de entender la evolución de la epidemia del coronavirus y poder predecir su evolución. Esta iniciativa está liderada por Iñaki Comas, investigador del Instituto de Biomedicina de Valencia (IBV-CSIC), y Mireia Coscolla, viróloga del Instituto de Biología Integrativa de Sistemas (I2SysBio) del CSIC y la Universidad de Valencia; junto con Fernando González Candelas, responsable de la Unidad Mixta de Investigación en Infección y Salud Pública de la Fundación para el Fomento de la Investigación Sanitaria y Biomédica (FISABIO) de la Generalitat Valenciana.
El trabajo de SeqCovid, junto con el de otras instituciones científicas del Estado, ha situado a España como el segundo país europeo y el cuarto del mundo en número de secuencias genómicas del virus obtenidas: un total de 4.244 secuencias, de las que 3.800 han sido generadas por el consorcio. Los datos obtenidos son depositados en bases de datos públicas, como la plataforma global NextStrain, para que grupos de todo el mundo puedan utilizarlas en sus análisis.
En su informe, SeqCovid muestra una fotografía genómica de la primera ola del SARS-CoV-2 en España, obtenida con el estudio de las secuencias de virus procedentes de 2.170 pacientes de todo el país. Las variantes del coronavirus se pueden diferenciar entre sí por las mutaciones que acumulan en sus genomas.
En la fotografía se observa que España presentó una diversidad única del virus en comparación con el resto de países de la Unión Europea. Ello se debe a la introducción temprana de linajes del virus. El inicio de la epidemia en nuestro país estuvo dominado por linajes A, descendientes directos de los primeros que aparecieron en China, mientras que en otros países la epidemia fue producida por linajes de aparición más tardía.
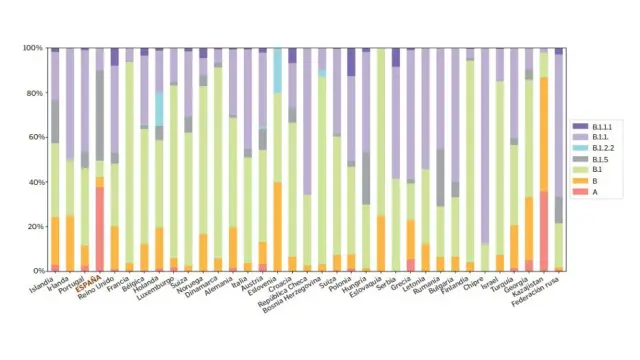
En España (cuarta columna) casi el 40% de las secuencias pertenecen a linajes A
Sin paciente cero
El caso más antiguo de covid-19 documentado en España es el de un hombre que regresó el 30 de enero de un viaje a Nepal y falleció, dos semanas más tarde, a causa de una neumonía de origen desconocido. Este caso temprano no implica que el paciente fuera el origen de la epidemia en España. Ni los estudios epidemiológicos, ni el análisis del consorcio, indican que el paciente generara ningún caso secundario, por lo que no existió un paciente cero.
Los datos filogenómicos (de parentesco genético entre las distintas muestras de virus secuenciadas) indican que el virus entró múltiples veces de manera independiente en España, como se ha observado en otros países. El estudio ha determinado que se produjeron un mínimo de 519 introducciones del virus antes del confinamiento. Los investigadores indican que este número debe tomarse con cautela porque representa el valor mínimo: el número de secuencias analizadas es mucho menor que el número de casos registrados en las primeras etapas de la epidemia. La mayor parte de las introducciones se produjeron desde mediados de febrero y hasta el estado de alarma.
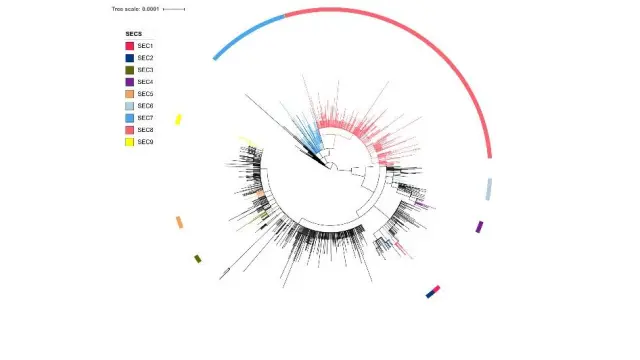
Unos pocos genotipos del virus generaron en España un gran número de casos secundarios. Predominan dos SEC –así llaman los investigadores a los clados o clases epidémicas españolas, es decir, las agrupaciones de casos producidos por un mismo genotipo del virus–. En la gráfica aparecen las muestras de origen español coloreadas según el SEC al que pertenecen. SEC8 y SEC7 reúnen al 30% y al 10%, respectivamente, de todas las muestras españolas. Esto indica que la introducción de dos genotipos concretos explica una parte importante de toda la epidemia en nuestro país. Además, los SEC8 y SEC7 son parte de los linajes A. Esto sugiere que las introducciones se dieron muy pronto en España, pues los linajes A fueron los primeros en aparecer en Asia, mientras que en Europa predominan los linajes B.
Nueve genotipos
El estudio también muestra que unos pocos genotipos del virus generaron un gran número de casos secundarios. Durante la primera ola, hasta nueve genotipos distintos tuvieron éxito y son los preponderantes entre las 2.170 muestras analizadas. Los autores denominan clados epidémicos o ‘clases epidémicas españolas’ (SEC por sus siglas en inglés) a las agrupaciones de casos producidos por un mismo genotipo del virus. Una parte considerable de la epidemia inicial se debió a la introducción de dos genotipos en concreto, SEC8 y SEC7, que forman parte de los linajes A, y que representan el 30% y el 10% respectivamente de todos los casos secuenciados. Son las dos clases epidémicas españolas que concentran el mayor número de casos y, por lo tanto, son los mayores grupos de dispersión en nuestro país.
Los datos también muestran el impacto que tuvieron las grandes ciudades en la dispersión del virus sobre su área de influencia. Si las ciudades hubieran actuado como centros dispersores del virus hacia zonas periféricas, deberíamos encontrar una mayor diversidad del virus en ellas y en las áreas metropolitanas que va disminuyendo a medida que nos alejamos. Los investigadores han podido confirmarlo estudiando el caso de Valencia y de otras ciudades como Gandía o Castellón.
El estudio sigue la evolución de los dos genotipos prevalentes y muestra que su dispersión disminuyó a mediados de marzo, lo que indica que el confinamiento fue muy eficiente y ayudó a evitar un número mucho mayor de casos y muertes.
Efecto fundador
Aunque determinar a qué se debe el éxito de unas variantes del virus frente a otras puede resultar complejo, los investigadores afirman que, en el inicio de una epidemia, raramente una mutación del virus se asocia a un mayor éxito epidemiológico o a una mayor virulencia. Es más probable que sea un proceso azaroso, en el que interviene lo que se denomina un efecto fundador. Los datos indican que los genotipos que se introdujeron antes –especialmente antes de que se tomaran las primeras medidas de contención– fueron los que más casos causaron. SEC8, por ejemplo, llegó a representar el 60% de los casos durante las primeras semanas de marzo.
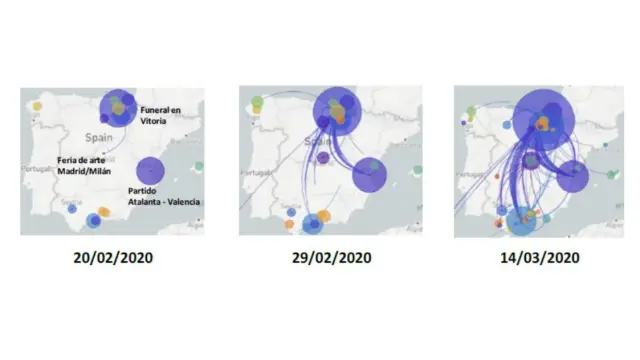
Eventos relacionados con el éxito de propagación de la variante SEC8SeqCovid
SEC8 llegó primero
Si el estudio genómico se cruza con los datos epidemiológicos, podemos tener más pistas de lo que sucedió. El genotipo dominante del virus en España, SEC8, se introdujo múltiples veces simultáneamente desde Italia durante la segunda quincena de febrero, al menos por dos vías, Madrid y Valencia. En Valencia fueron probablemente los aficionados que asistieron al partido de fútbol de la Liga de Campeones entre el Atalanta y el Valencia, el 19 de febrero en Milán, quienes lo introdujeron. En Milán se celebró esos días una feria de moda, y poco después tuvo lugar una feria de arte en Madrid. Existe evidencia de que algunos de los casos importados de Italia a través de Valencia estuvieron en sitios públicos en Madrid. También hubo brotes tempranos de SEC8 en el País Vasco/La Rioja y en Andalucía, pero no se sabe si fueron introducciones independientes del virus desde Italia o a partir de casos de Valencia y Madrid.
Probablemente se produjeron distintos eventos de superdispersión local junto con una movilidad posterior que ayudó a extender el SEC8, que en poco tiempo dominó la epidemia en España. Un ejemplo de evento de superdispersión fue un funeral celebrado en Vitoria el 28 de febrero. La entrada de otros genotipos se sitúa entre finales del mes de febrero y el mes de marzo, representando brotes grandes pero localizados. Los datos del SeqCovid demuestran que, en la primera ola, los eventos de superdispersión tuvieron un gran impacto en el éxito de la epidemia.
Un análisis preliminar de los datos de la segunda ola indica una presencia muy residual de los nueve genotipos que dominaron la primera ola. Prácticamente todos se han extinguido, lo que demuestra el gran éxito que representó el confinamiento.
20A.EU1, la variante que domina la segunda ola, apareció en Aragón
Un reciente estudio internacional en el que han participado la Universidad de Basilea, el ETH Zürich y el consorcio SeqCovid ha identificado una nueva variante del SARS-CoV-2, bautizada como 20A.EU1, que domina la segunda ola de la pandemia en España. Esta variante apareció hacia finales de junio en Aragón, en Alcañiz y Caspe, en un evento de superdifusión entre trabajadores agrícolas, y rápidamente pasó a Cataluña. A partir de aquí, se transmitió a la población local y continuó su rápida expansión por todo el país hasta llegar a representar casi el 80% de las secuencias del virus en España.
No hay datos que indiquen que esta variante del virus sea más contagiosa o virulenta. Su éxito se debe probablemente a que el primer evento de superdifusión que se dio entre temporeros desprotegidos, en Aragón y Cataluña, coincidió con la relajación de las restricciones de viaje y de las medidas de distanciamiento social. El estudio pone de manifiesto la importancia de las medidas de control de la movilidad para evitar la expansión del virus. La reanudación del turismo en verano en nuestro país ha facilitado que la variante 20A.EU1 se haya propagado por toda Europa, donde se ha identificado en al menos 12 países: representa el 90% de los casos del Reino Unido, el 60% de Irlanda y un 30-40% en Suiza y los Países Bajos. Incluso se ha detectado en lugares remotos como Hong Kong, Australia o Nueva Zelanda.
-Ir al suplemento Tercer Milenio
Apúntate y recibe cada semana en tu correo la newsletter de Tercer Milenio